- 网页首站
- 企业展示
- 产品中心
- 新闻动态
- 合作伙伴
- 招贤纳士
- 联系我们
在过去的十年中, RNA-seq 已经成为全转录组范围内分析差异基因表达和 mRNAs 差异剪接的重要工具。然而,随着下一代测序技术的发展,RNA-seq 技术也在不断发展。RNA-seq 可用于研究 RNA 生物学的方方面面,包括基因表达、翻译(翻译组,translatome)、RNA 结构(结构组,structurome)、调节性 RNA、RNA 表观遗传学以及 RNA 动力学等等。RNA-seq 的其它应用也在开发中,例如空间转录学 (spatialomics)。加上近年来长读长测序和直接 RNA-seq(direct RNA-seq) 技术的应用以及数据分析计算工具更好的整合,RNA-seq 技术的创新使人们对 RNA 生物学有更全面地理解,例如从何时何地转录发生到控制 RNA 功能的折叠和分子间相互作用等问题。
回顾 RNA-seq 技术的整个十年,自其诞生之日起,RNA-seq 就成了研究分子生物学的普遍工具,这项技术几乎构成了我们对基因组功能的认知基础。RNA-seq 中常用的分析方法就是找出差异基因表达 (Differential gene expression, DGE)。从始至今,DGE 分析从未发生实质性的改变。虽然 RNA-seq 这个术语经常被用于那些完全不同的方法学方法和/或生物学,但是 DGE 分析仍然是 RNA-seq 主要的应用场景,并被视为常规研究工具。
科学研究的突破通常都是基于技术手段的突破更新,在 RNAseq 之前,基因芯片技术是基因组表达分析的中坚力量,在这个技术辉煌的时期,一提到基因表达研究,人们就会想到使用芯片。芯片技术的简单、方便、快捷,能较准确的找到研究者需要找到的基因组信息使它曾风靡一时。然而与 RNAseq 相比,芯片技术的局限性就显而易见了。1)芯片技术须依赖已知的基因组信息,在探索性研究和非模式生物的研究中存在局限性;RNAseq 在这方面的优势明显,它不需要预先设计探针,因此得到的数据集是无偏倚的,实现了无假设的实验设计 1,2,对未知转录本和变异发现研究而言提供了强有力的工具,这点芯片是无法实现的。2)芯片技术动态范围较窄和灵敏度低,芯片技术的动态范围通常为 3 个数量级(103),而 RNAseq 的动态范围要比芯片技术高几个数量级,可跨越 5 个数量级(>105)1,3,因此,RNAseq 可检测的差异表达基因比例要高于芯片,特别是低丰度的基因 3,4。3) 芯片技术通常无法完成对选择性剪接位点和新型异构体以及非编码 RNA 的检测,而 RNA-Seq 除了基因表达谱分析,还能鉴定选择性剪接异构体、剪接位点和等位基因特异的表达–所有这些都在单个实验中完成。因此,RNAseq 的出现很快就成为了 RNA 研究的主流手段。
近十年来 RNAseq 技术也是不断的发展,至今,从标准的 RNAseq 方法中延申出多达 100 种的 RNAseq 方法。在 RNA 的文库构建上也有了改良,如发表在 Nature Methods 上的以 mRNA 3』端序列构建文库的方法,其采用的是 Lexogen 公司的 Quantseq 文库构建试剂盒 5。其中以 Illumina 为典型的短读长测序平台能对这些由大部分不同方法的 RNAseq 构建的文库进行测序,另外,近几年长读长 RNAseq 以及直接 RNAseq 的进步弥补了短序列测序无法解决的问题,如在基因融合,结构变异,以及不同剪切体上有更加深入的了解。
那么,我们来看看常规 RNAseq 与 3’mRNAseq 之间各自的优劣势,以便我们在今后的研究中选择自己更有利的方法进行研究。
RNA-seq VS Quantseq
流程:常规 RNAseq VS Quantseq
常规 RNAseq 我们以典型的 Illumina truseq RNA 建库试剂盒为例与 Quantseq 3』 mRNA 建库试剂盒进行比较。在常规的 RNAseq 测序流程中,通常会包含以下几个步骤, mRNA 富集或去除核糖体 RNA(这里需要跟 RNA 的质量以及研究方向进行相应选择,对于降解的 RNA 样品,如 RIN<7 的样品应选择去除核糖体 RNA),RNA 片段化,第一链 cDNA 合成,第二链 cDNA 合成,加 A 尾加接头,文库扩增这些步骤,当然中间会有纯化的步骤(Fig.1 左)。在常规 RNAseq 中,通常需要 1ng-2 μg 的 total RNA input 量。然而 QuantSeq 通常比标准 RNA-seq 法流程更为简单且需更低的 total RNA input 量:100pg-1 μg。在 Quantseq 流程中,每个转录本只产生一个片段,因此数据量是常规 RNAseq 的 1/10,并可以配合 UMI 模块对第二链 cDNA 进行单分子标签标记,使基因表达定量更加精确。QuantSeq 采用 oligo dT 引物特异逆转录含 poly(A) 尾 mRNA,第二链使用随机引物进行合成,随机引物结合的位置与 poly(A)之间的距离决定了插入片段的长度,因此不需要 poly(A) 富集及片段化这一步骤,并在 cDNA 合成后立即进行 PCR,从而取代了接头连接步骤使整个建库流程时间大大缩短。这种方法可以在低测序深度上实现与标准 RNA-seq 同等的灵敏度水平。由于 Quantseq 中每个样本需要的数据量小,因此,这种方法可以实现更多个文库的混合同步测序(Fig.2 中和右)。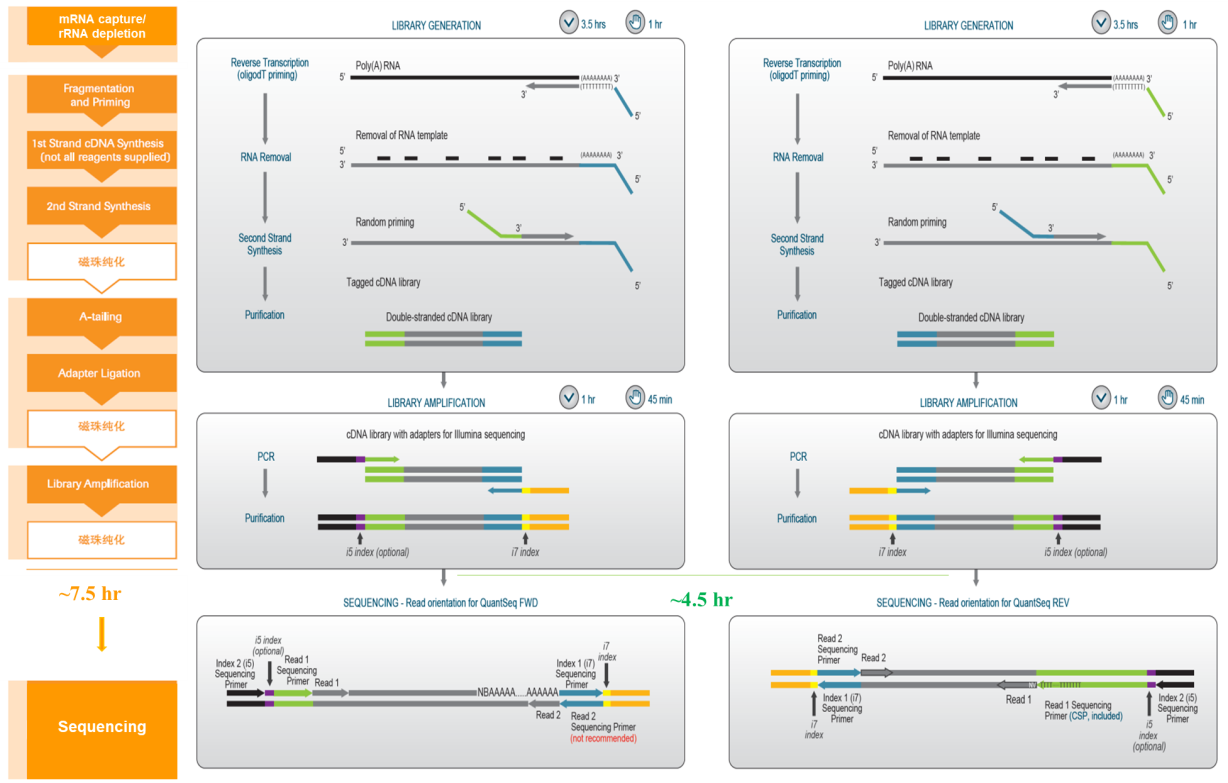
Fig.1 左边:为常规 RNAseq 建库测序流程,中间和右边分别为 quantseq 3』 mRNA FWD 和 REV 建库测序流程。中间:QuantSeq FWD 试剂盒 Read 1(从绿色 P5 接头部分开始)测序对应的是靠近 mRNA 3』端序列,可以使用 Illumina 测序引物进行测序,且费用较低。右边:QuantSeq REV 试剂盒测序位置是 Read 1 和 Read 2 的互换,Read 1 能够直接检测到转录本的末端。QuantSeq REV Read 1 测序需要定制化的测序引物(CSP, 包含在试剂盒内)。
应用方向: 常规 RNAseq VS Quantseq
在常规 RNAseq 应用中主是以 DGE 分析为主,通常每个样本会测 20-30 M 的 reads 数进行高质量的 DGE 分析。此外,由于常规 RNAseq 对整个转录本的序列进行打断后测序,其覆盖了转录本的完整信息(Fig. 4),因此除了主要的 DGE 分析外,它可以进行转录本的 de novo 组装,Isoform 的检测、定量以及基因融合的分析(Fig.2)。对于后几项的应用,它们对数据量上有很大的提升要求,如 Isoform 检测需 70-80M 的 reads 每个样本 6,全转录组则需 100M 的 reads 数每个样本 7(Fig.3)。相对于 Quantseq 3’mRNA seq,其富集的是 mRNA3』 CDs 以及 UTR 区域 (Fig.4),每个转录本只产生一个片段 (Fig.2),因此,仅需很少的数据量就可以进行准确的 DGE 分析,通常为 3-10M 的 reads 数 8,仅为常规 RNAseq 的 1/10(Fig. 2 和 3),因此大大节省测序空间,允许更多样品的混合测序,大大节省了成本。此外,mRNAs 的 APA 化会产生 3ʹ UTR 长度不等的异构体。对于一个特定的基因来说,它不仅产生了这个基因的多个亚型,而且由于 3ʹUTR 中存在着顺式调控元件,这也会影响该转录本的调控。因此,Quantseq 对 APA 的研究者们来说可用于更详细地研究 miRNA 的调控作用,mRNA 的稳定和定位,以及 mRNA 的翻译。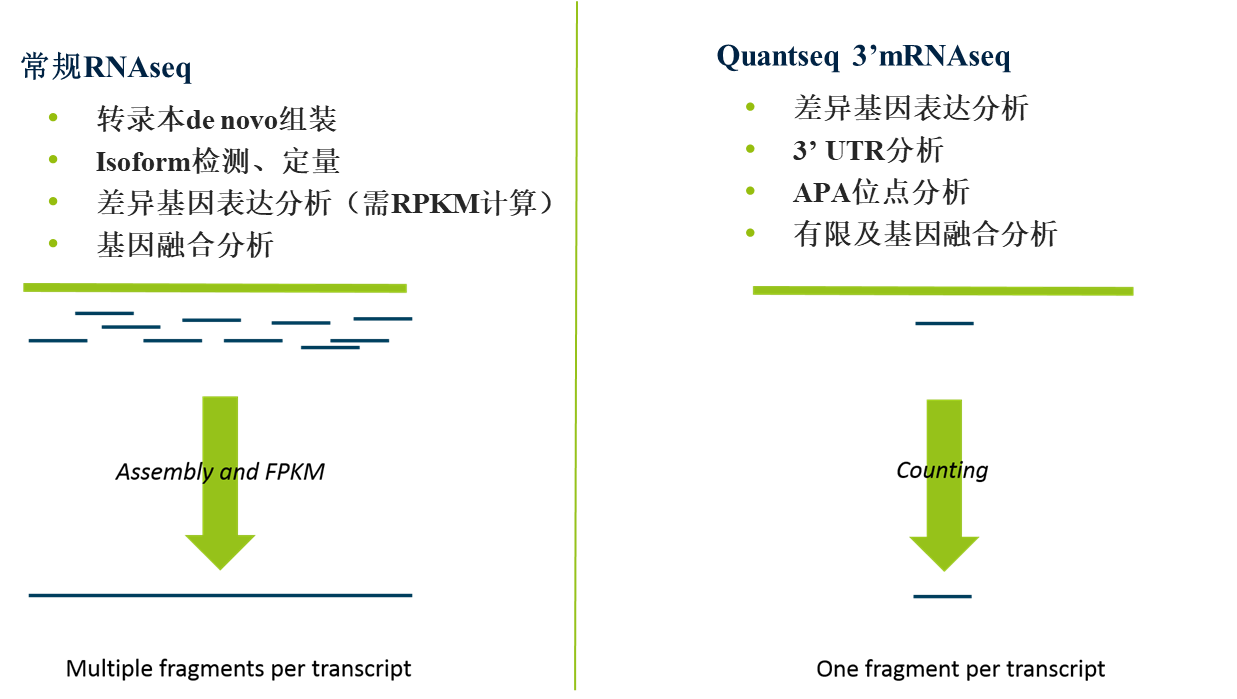
Fig.2 常规 RNAseq 与 quantseq 3』 mRNAseq 在应用上的区别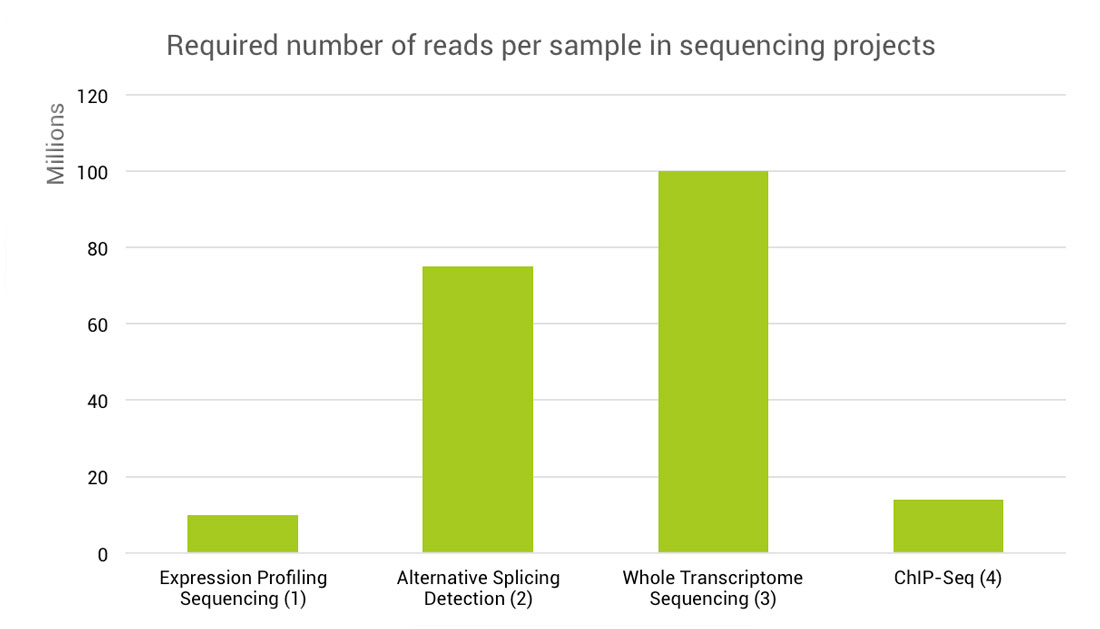
Fig.3 不同应用对数据量上的要求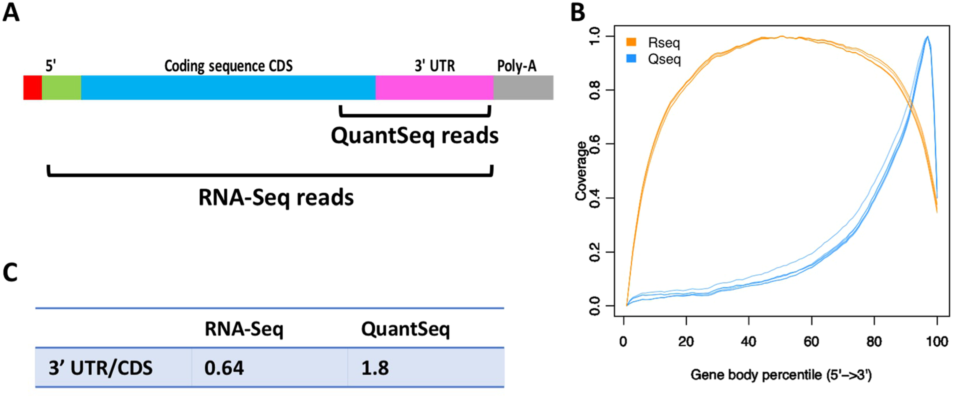
Fig.4 常规 RNAseq 与 Quantseq 3』 mRNA seq reads 在转录本的覆盖情况。
对于 isoform 检测、定量研究,短读长的建库方案及测序平台存在着较大的缺陷 9。Weirather JL 等人 9 通过采用金标准的 Spike-in RNA 标准品 SIRVs E0(Lexogen)对 PacBio, ONT 以及 Illumina 测序平台对基因 isoform 检测性能进行了评估。E0 模块包含来自 7 个人类模型基因的 69 种 isoform 等摩尔比例组成,综合反映了可变剪接、可变转录起始和终止位点、重叠基因和反义转录的变化(Fig.5)。Weirather JL 等人采用 Lexogen 提供的 3 种注释文库对不同测序平台进行了 isoform 检测性能评估,分别为「correct library」,注释包含所有 68 种真实表达的 isoforms; 「insufficient library」,注释仅包含 68 种真正表达 isoforms 中的 43 种; 以及「over-annotated library」,注释含 68 个真实表达的 isoforms 和额外 32 个未表达的 isoforms。3 个注释文库,Illumina 数据经 StringTie 进行 isoforms 重构,分别检测到 44,63,62,其中分别有 33,27,24 假阳性预测; ONT 测序直接检测到 correct library 文库中所有 68 个表达 isoforms(Table 1),PacBio 测序检测到 67 个,其中一个 219bp isoform SIRV618 因片段筛选时已过滤掉了。因此,相对 Illumina,PacBio 和 ONT 在 isoforms 检测中表现出超高优势。同时表明短读长拼接重构 isoforms 存在较大的缺陷,因此,常规 RNAseq 较 Quantseq 在 isoforms 检测中的优势也就显得没什么意义了。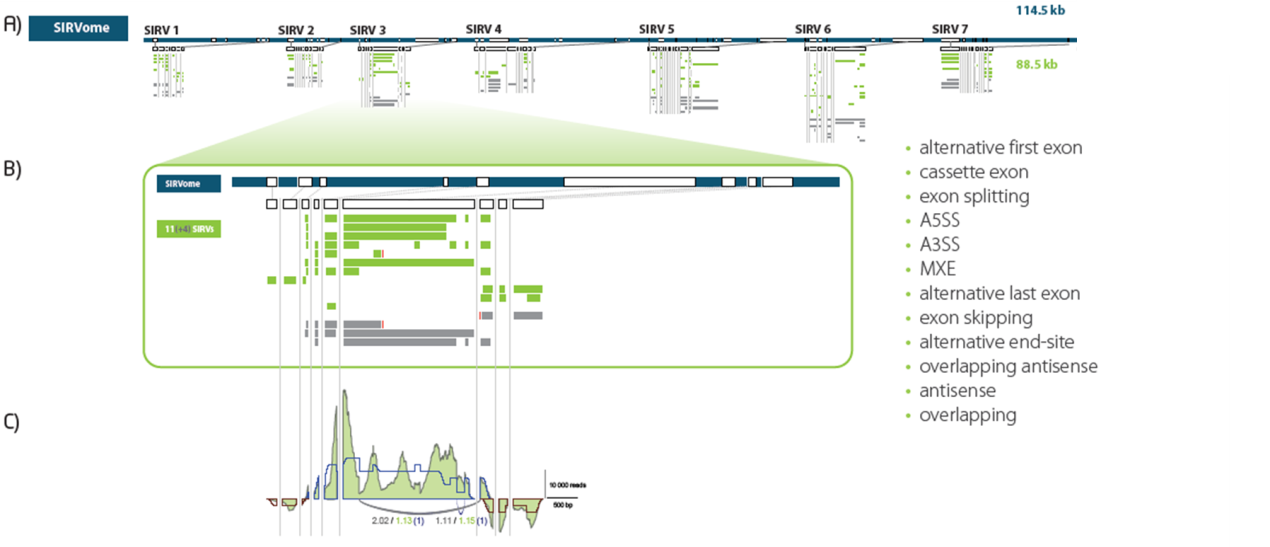
Fig.5 SIRV 设计概览。 SIRV1 到 SIRV7,模拟人类模型基因,全面代表了主要的可变剪切方式以及重复和差异转录。A)7 个 SIRV 基因的人工染色体,即 SIRVome;B)SIRV3 的放大图,提供 11 个转录本可变剪切体(绿色);灰色区域的转录本可变剪切体是附加的注释,用于其他的评估程序;C)SIRV mix 中已知的转录本 isoform 浓度可以与预期的基因和外显子连接覆盖范围 (蓝线为正链,红线为负链) 与实验获得的 reads 覆盖范围 (绿色区域) 进行比较。
Table 1. Illumina、PacBio 和 ONT 在金标准 SIRVs 中 isoform 鉴定的表现。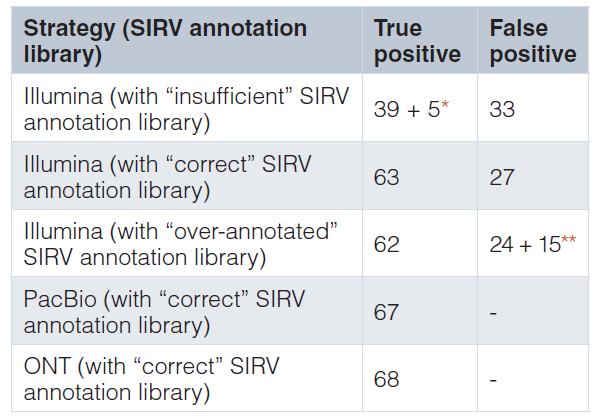
* 在「insufficient 」的 SIRV 注释文库,其中有 25 种 isoforms 未被注释但表达。在这 25 个 isoforms 中,有 5 个 isoforms 在 Illumina 测序平台被检测到。
** 在「over-annotated」的 SIRV 注释文库中,包含额外注释的 32 种 isoforms,但没有真正表达。在这 32 种未表达的 isoforms 中,共检测到其中 15 个 isoforms。
定量精确度:常规 RNAseq VS Quantseq
Moll, P. 等人用 ERCC spike-in RNA 标准品的转录本覆盖 reads 数与 input 的分子数作图进行分析 (Fig. 6)。在线性模型评估和 Spearman 关联性评估中,QuantSeq 展现出了非常高的 input-output 关联性和基因表达测定的准确性 9。同时, 通过使用「erccdash- board」 软件,对 QuantSeq 和常规 mRNA-Seq 的差异基因表达检测能力进行了对比。当测定的读数 (Reads) 由 10M 降低到 0.625M,QuantSeq 维持了相当高的曲线面积值(AUC 0.860-0.897),而 mRNA-Seq 的曲线面积值较低,在 0.736 到 0.776 之间 (Fig. 7)9。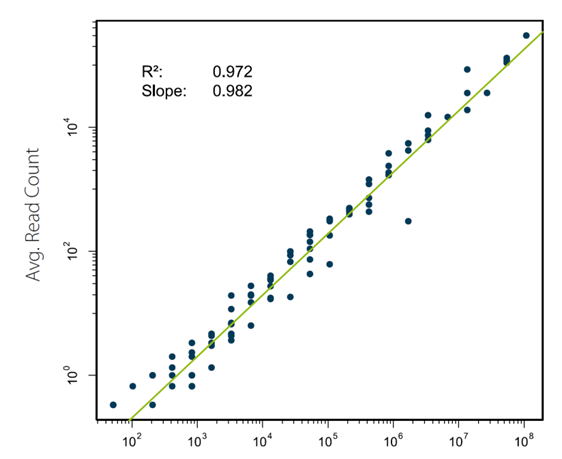
Fig. 6 QuantSeq-源于 ERCC reads 数与给定的 input 间存在极好的关联性。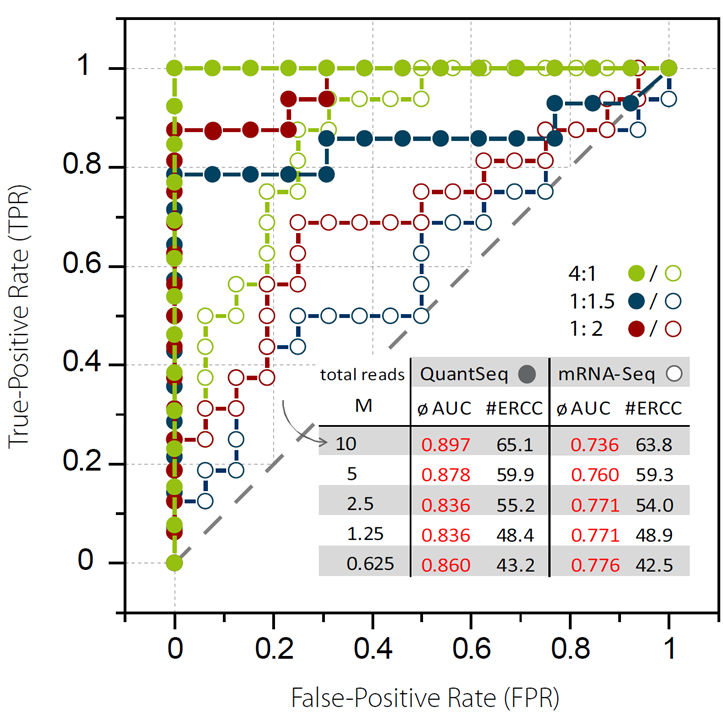
Fig. 7 QuantSeq 和常规 mRNA-Seq 基因差异表达分析。给定的 ERCC ExFold Spike-In Mix1 和 Mix 2 间的倍数变化(4:1,1:1.5,1:2)用于评估真假阳性率 (TPRs 和 FPRs优的基因差异表达检测体现在值为 1 的曲线面积(AUC)。用 AUC 对 ERCC 检测到的 RNA reads 进行评估(测序数据量从 10 M 降到 0.625 M)。
低质量 RNA 中的表现:常规 RNAseq VS Quantseq
对于低质量,降解的 RNA 样本,如 FFPE 样本,常规的 mRNA 建库会导致 3』端的偏好性,因此通常需要 rRNA 去除之后在进行建库,这样大大提高了建库的成本。而 Quantseq 3’mRMA 文库构建本身就是以 mRNA 的 3』端序列进行文库构建,主要集中在转录本的 3』端,每个转录本只产生 1 个片段。这样能够使得无论 RNA 的质量如何(包含 FFPE 样本)都可准确定量。因此,相对于其他用 Poly(A)分选 mRNA 操作流程,Quantseq 3’mRNA-Seq 更有效的对低质量的样本进行建库。
通过使用同一来源的不同质量 RNA 样本进行比较,评估 Quantseq 适用于高度降解的样本(如 FFPE 样本)的能力。将人的 MOLP-8 肿瘤细胞系分成两份,一份进行新鲜冷冻,一份处理成 FFPE 样本,从而使同一个来源的样本得到不同质量的 RNA。用 RIN 值(RNA 完整度)来区分 RNA 的质量。RIN 值大于 8 表示 RNA 质量高。对应严重降解的样本,RIN 值不适用于质量的评估,因此使用 DV200 值(大于 200nt 的 RNA 片段的分布值)来表示 RNA 质量。低完整度的 RNA 对应低 DV200 值。RNA 提取后,FFPE 样本的 DV200 值为 87%(RIN 值 2.8),冷冻样本 RIN 值为 8.3。 使用 50ng 总 RNA,用 QuantSeq FWD 试剂盒进行文库构建。FFPE 样本提取的 RNA,即使 DV200 值低至 23%(数据未显示)仍能成功构建 Quantseq 文库。文库在 Hiseq2500 上进行用 1x 50 bp 读长测序。结果显示 FFPE-RNA 文库和冷冻保存 RNA 文库的基因表达的相关性很高(R²= 0.86),表示 Quantseq 能在不同质量的 RNA 中表现稳定(Fig.8)。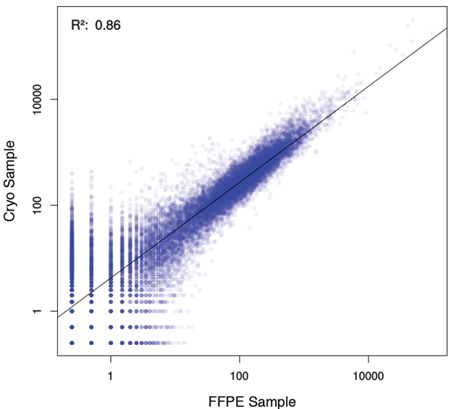
Fig.8 FFPE 和冷冻样本的基因表达的相关性。
数据分析:常规 RNAseq VS Quantseq
常规的 RNAseq 是以打断后的转录本进行建库测序,其覆盖的是整个转录本,因此与 Quantseq 每个转录本只生成一个片段相比,其在数据上要远远大 Quantseq 的数据量;另外,常规 RNAseq 的数据在分析时需要将打断的转录本进行拼接以及 RPKM 的计算等流程,而 Quantseq 无需复杂的计算,仅需基因片段的计数即可获得基因表达数据。因此,前者需要耗费大量的计算资源以及计算时间,而 Quantseq 的数据分析就显得非常简单而省时了(6 个样品 35 min 内即可完成分析 9,现在会更快)。这对于讲究时效性的基于基因表达变化的辅助诊断及治疗来说是非常有利的。
此外,Lexogen 与 Bluebee® Genomics Platform 达成战略合作,为 QuantSeq 3』 mRNA-Seq 建库试剂盒 (FWD 和 REV) 的数据提供免费分析。Lexogen 为每个试剂盒提供一个密码,使用者可以登录该平台分析数据。也可用 Partek Flow (license required) 平台进行数据分析。使用者可以直接在 Partek Flow 导入原始测序数据,然后进行自动化分析。也可进行定制化分析。
技术在不断更迭,在诸多的技术手段中找到适合自己的研究往往就是成功的开始。QuantSeq 3』 mRNA-Seq 在诸多方面都有良好的体现,特别在基因表达分析,3』UTR 以及 APA 位点的分析等其他方面优势明显,因此,在关注于以上几点的 RNA 测序研究应用中,QuantSeq 3』 mRNA-Seq 是可谓是不二之选。
Lexogen 是一家专注于为 RNA 研究提供创新性解决方案的生物公司,产品线覆盖全面,从样本制备,RNA 提取,RNA 文库构建到最后的数据分析,Lexogen 可提供完整的解决方案。其独特的链特异性文库构建流程,无需 RNA 打断,低 input 量要求,流程简单快速,仅需 4.5 h 即可完成文库的构建,可大大节约时间成本。北京仲黎商贸有限公司作为贝克曼库尔特(中国)的战略合作代理商,同时也是 Lexogen 公司在中国的代理商,我们将竭尽所能为您提供优质的服务,欢迎咨询 sales@bjzltrade.com!
1. Wang Z, Gerstein M, Snyder M. RNA‑Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 2009;10:57‑63.
2. Wilhelm BT, Landry JR. RNA‑Seq—quantitative measurement of expression through massively parallel RNA‑Sequencing. Methods. 2009;48:249‑57.
3. Zhao S, Fung-Leung WP, Bittner A, Ngo K, Liu X. Comparison of RNA‑Seq and microarray in transcriptome profiling of activated T cells. PLoS One. 2014;16;9(1):e78644.
4. Wang C, Gong B, Bushel PR, et al. The concordance between RNA-seq and microarray data depends on chemical treatment and transcript abundance. Nat Biotechnol. 2014;32(9):926‐932. doi:10.1038/nbt.3001.
5. Moll, P., Ante, M., Seitz, A. et al. QuantSeq 3′ mRNA sequencing for RNA quantification. Nat Methods 11, i–iii (2014).
6. Liu Y., et al., Evaluating the impact of sequencing depth on transcriptome profiling in human adipose. Plos One 8(6):e66883 (2013)
7. Bentley, D. R. et al. Accurate whole human genome sequencing using reversible terminator chemistry. Nature 456, 53–59 (2008)
8. Liu Y., et al., RNA-seq differential expression studies: more sequence or more replication? Bioinformatics 30(3):301-304 (2014)
9. Weirather JL, de Cesare M, Wang Y, Piazza P, Sebastiano V, Wang XJ, Buck D, Au KF. Comprehensive comparison of Pacific Biosciences and Oxford Nanopore Technologies and their applications to transcriptome analysis. F1000Res. 2017; 6 100. doi:10.12688/f1000research.10571.2. PMID: 28868132; PMCID: PMC5553090.