- 网页首站
- 企业展示
- 产品中心
- 新闻动态
- 合作伙伴
- 招贤纳士
- 联系我们
Quant-seq表达谱文库构建试剂盒


QuantSeq 3' mRNA-seq文库构建试剂盒是以mRNA靠近3' 端序列进行文库构建,文库可兼容Illumina和Ion Torrent测序平台。每个转录本只产生一个片段,因此能准确进行基因表达定量,同时减少测序深度,能使更多样本混样测序。
产品优势
快速简单的一体化实验方案: 从总RNA到可用于测序的文库时间不超过4.5小时;
每个转录本仅有一个片段产生—可进行全基因组范围的基因表达分析;
能稳定、灵敏检测低丰度的转录本;
适用于低起始量(100pg(FWD)或10ng(REV)总RNA)及低质量RNA(如FFPE样本);
可高效替代芯片检测或传统的RNA测序,友好匹配自动化工作站;
在Blubee基因组分析平台上免费进行数据分析;
NEW! 节约成本,优化的globin mRNA去除流程可用于QuantSeq文库构建;Dual indexes能支持9,216个 i5/i7 index,或者可选择96个unique index,同时提供能够检测、排除扩增偏好性的UMIs。
建库流程
QuantSeq 文库构建试剂盒通过oligo dT引物特异逆转录含poly (A)尾的mRNA,第二链使用随机引物进行合成。由于插入片段的大小是由第二链合成引物和poly(A)尾之间的距离决定的,因此无需额外进行 RNA 片段化。随后的PCR可以提供9,216个不同的i5/i7index组合用于Illumina平台,48个index 用于Ion Torrent平台。能够实现更多样本混样测序。
Lexogen提供两个版本的Illumina Quantseq 3' mRNA-Seq文库构建试剂盒,不同处在于Illumina接头序列的位置。下图绿色的是Read 1的序列,蓝色的是Read 2的序列。Quant-Seq Forward (FWD) 在第二链合成的引物中包含Read 1序列(Fig. 1, 左)。因此,测序的方向是朝向poly(A)尾的方向。推荐使用这种方法进行基因表达的分析。如果研究关注的是转录的3’端序列或者双端测序策略,推荐使用QuantSeq Reverse (REV),Read 1 接头序列由oligo-(dT)引物引入(Fig. 1, 右)。
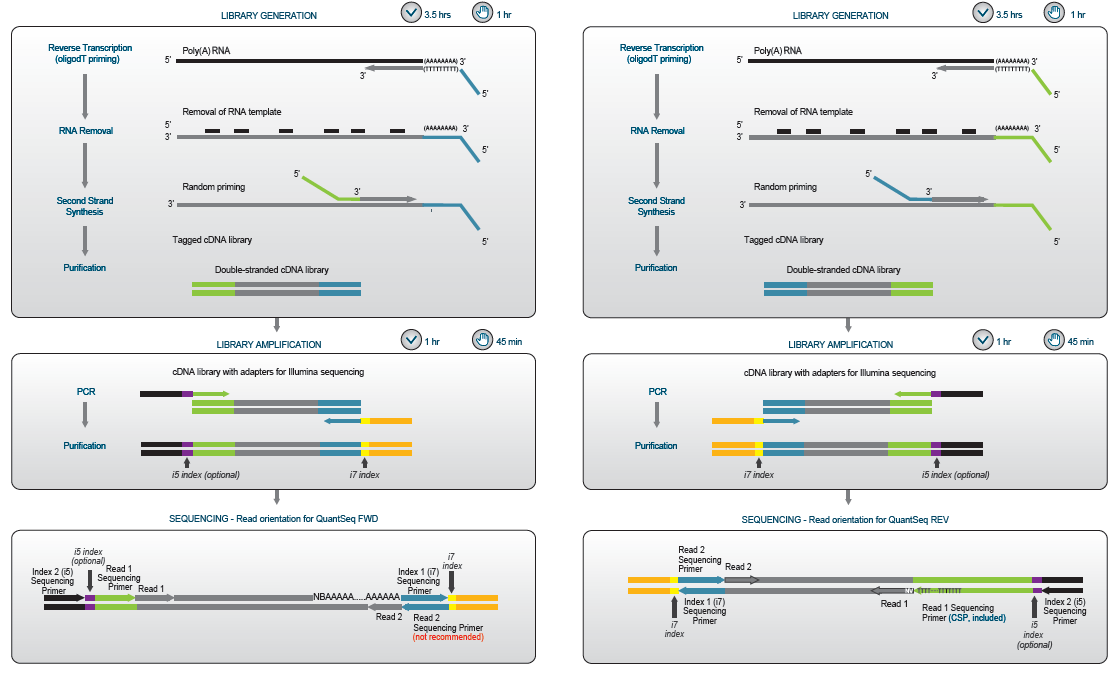
Figure 1 | QuantSeq 3' mRNA-Seq FWD 文库构建试剂盒(左图) 和 QuantSeq 3' mRNA-Seq REV文库构建试剂盒 (右图) 建库流程图。
左边:QuantSeq FWD试剂盒Read 1(从绿色P5接头部分开始)测序对应的是靠近mRNA 3' 端序列,可以使用Illumina测序引物进行测序,且费用较低。右边:QuantSeq REV 试剂盒测序位置是 Read 1 和Read 2 的互换,Read 1 能够直接检测到转录本的末端。QuantSeq REV Read 1测序需要定制化的测序引物(CSP,包含在试剂盒内)。
性能表现
3’ 端链特异性比对以FDA提供的测序质控 (SEQC) 标准样品A和B为样本(A和B分别是ERCC外源RNA与有参RNA的混合物1和混合物2)1,2,用QuantSeq试剂盒进行文库构建,测序数据与近期生物分子资源实验室协会 (ABRF) 发布的NGS研究中的mRNA-Seq数据组比对3。在常规mRNASeq中,测序Reads覆盖转录本的全长,而QuantSeq的Reads仅覆盖转录本的3’端(Fig.2)。QuantSeq在精确检测基因表达水平的同时,节省了大于90%的测序深度。由于spike-in转录本(RNA质控品)仅来源于正义链的转录,因此链特异性的评估可以不依赖于于任何基因组注释。在各种情况下QuantSeq数据均显示出高于99.9%的链特异性,而两个被评估的mRNA-Seq SEQC数据集(data sets)链特异性仅分别为93.4%和97.8%3。QuantSeq可降低噪音,能够实现准确检测及定量反义转录本。
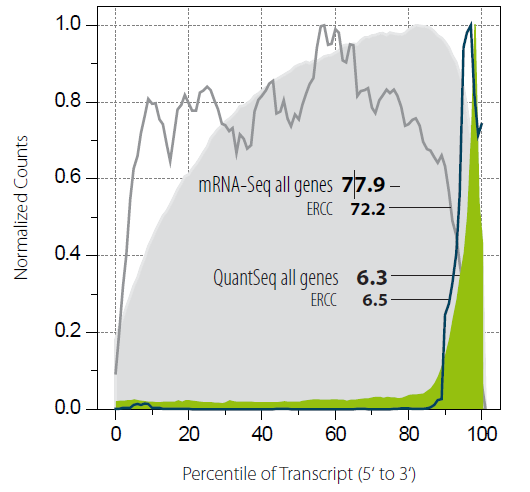
Figure 2 | QuantSeq和标准mRNA-seq的覆盖度与标准化转录本长度的对比。RSeQC是所有转录本(面积),ERCC混合物(线性,QuantSeq (彩色)和mRNA-Seq (灰色) )的覆盖度信息。曲线面积值(AUC)作为测序覆盖度的测量尺度。
QuantSeq量化范围宽--6个数量级
为了分析QuantSeq的基因计算准确性,用ERCC 的spike-in转录本覆盖reads数与input的分子数作图进行分析 (Fig.3)。在线性模型评估和Spearman关联性评估中,QuantSeq展现出了非常高的input-output关联性和基因表达测定的准确性。
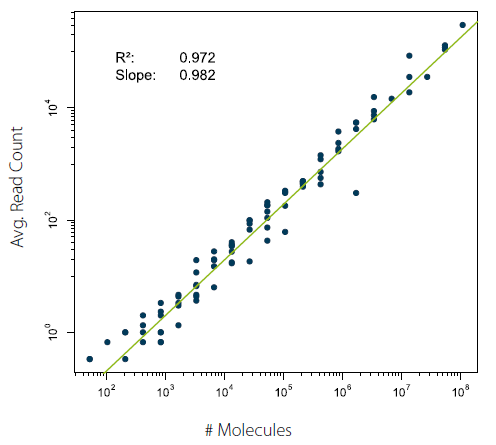
Figure 3 | QuantSeq 获得的ERCC reads数与给定的input间存在更好的关联性。
基因差异表达分析
通过使用“erccdash- board” 软件 4,对QuantSeq和常规mRNA-Seq的基因差异表达的检测能力进行了对比。当测序的Reads数由10M降低到0.625M,QuantSeq维持了相当高的曲线面积值(AUC 0.860-0.897),而常规mRNA-Seq的曲线面积值较低,在0.736到0.776之间(Fig. 4).
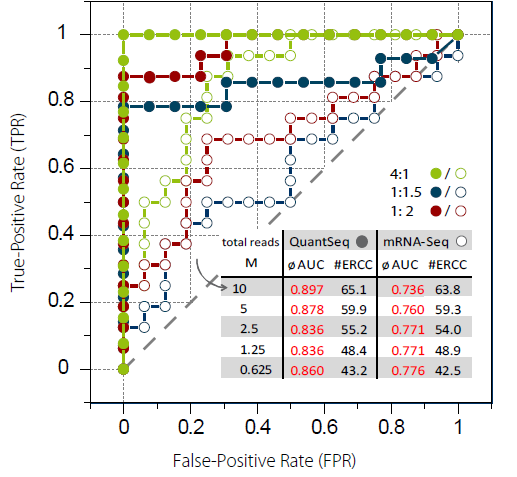
Figure 4 | QuantSeq和常规mRNA-Seq在基因差异表达分析上的性能表现。给定的ERCC ExFold Spike-In Mix1和Mix 2间的倍数变化(4:1 / , 1:1.5 / , 1:2 / ) 用于评估真假阳性率(TPRs 和FPRs)。优的基因差异表达检测体现在值为1的曲线面积(AUC)。用AUC对 检测到的ERCC RNA reads进行评估(测序数据量从10 M降到0.625 M)。
Quantseq在低质量RNA中的表现
Quantseq在高质量和低质量(FFPE)样本中有很好的相关性
用同一来源的不同质量RNA样本进行比较,评估Quantseq适用于高度降解的样本(如FFPE样本)的能力。
将人的MOLP-8肿瘤细胞系分成两份,一份进行新鲜冷冻,一份处理成FFPE样本,从而使同一个来源的样本得到不同质量的RNA。用RIN值(RNA完整度)来区分RNA的质量。RIN值大于8表示RNA质量高。对于严重降解的样本,RIN值不适用于质量的评估,因此使用DV200值(大于200 nt的RNA片段的分布值)来表示RNA质量,低完整度的RNA对应低DV200值。
RNA提取后,FFPE样本的DV200值为87% (RIN值2.8),冷冻样本RIN值为8.3。使用50ng总RNA,用QuantSeq FWD 试剂盒进行文库构建。FFPE样本提取的RNA,即使DV200值低至23%(数据未显示),Quan-tseq 仍能成功构建文库。FFPE样本有对应的低质量RNA建库操作流程,对应冷冻样本,应用标准操作流程。文库在Hiseq2500上进行用 1x 50 bp读长测序(Fig.5)。
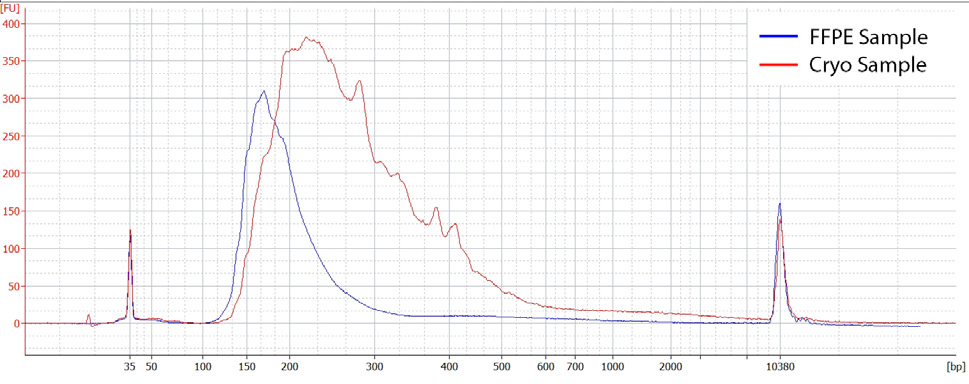
Figure 5 | 安捷伦2100 HS DNA芯片检测QuantSeq 3’mRNASeq FWD试剂盒构建的FFPE(蓝色)或冷冻样本(红色)的RNA文库。对于降解的样本来说,文库是一个较为平滑的分布,没有明显可见的锯齿突起,且整体片段偏小。平均片段长度204bp(FFPE)和286bp(冷冻样本)。
覆盖度与转录本的长度的相关性显示覆盖度关注于转录本的3’端,与RNA的质量无关(Fig.6)。但是,由于QuantSeq FWD文库对poly(A)端进行测序,覆盖度取决于文库的片段大小和测序读长。FFPE样本文库明显比冷冻样本文库短,因此,会更多的读取转录本的3’端,并反映在覆盖曲线中。
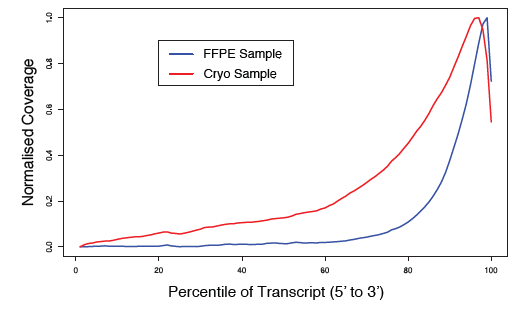
Figure 6 | 来源于FFPE-RNA(蓝色)和冷冻保存RNA(红色)的NGS文库的Quantseq read均一化覆盖度与百分位数转录本长度的相关性。
FFPE-RNA文库和冷冻保存RNA文库的基因表达的相关性很高(R² = 0.86),表示Quantseq能在不同质量的RNA中表现稳定(Fig.7)。
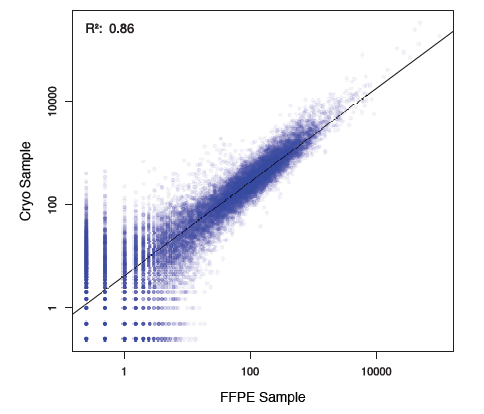
Figure 7 | FFPE和冷冻样本的基因表达的相关性。
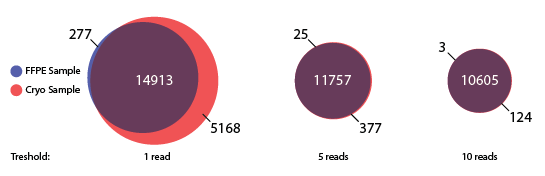
Figure 8 | QuantSeq在FFPE和冷冻样本中以2.5 M的统一测序深度检测到的基因的韦恩图(reads/基因的阈值分别为1, 5, 10)。
Quantseq有非常高的灵敏度。测序量在26.5M reads时, RNA完整性好的新鲜冷冻样本中检测到25,842个基因(数据未显示)。当单个样本测序数据量在2.5M时,在新鲜冷冻样本中检测出20,081个基因,且每个基因至少有1个read;相应的,在FFPE样本中能检测到15,190个,两者有24%的差异(Fig.8)。但是,当阈值提高到5或者10个reads/基因时,差异分别下降至3%和1%。这些比对结果表明,QuantSeq能够可靠地对冷冻保存和FFPE样品进行基因表达分析。两者之间在低表达基因检测存在的差异,是由于FFPE样本在处理、储存及回收过程中低拷贝转录本很容易降解至无法检测到。
Quantseq用oligo(dT)引物进行逆转录,每个转录本只产生1个片段。这样能够使得无论RNA的质量如何(包含FFPE样本)都可准确定量。常规的mRNA-seq操作流程旨在覆盖整个转录本,但是在使用降解样本时将导致严重的3’偏好性。因此,相对于其他用Poly(A)分选mRNA操作流程,Quantseq 3’mRNA-Seq更有效的对低质量的样本进行建库。
订购信息:
| Product # | Product | Kit Size |
| 015.24 | QuantSeq 3‘ mRNA-Seq Library Prep Kit (FWD) for Illumina, 24 preps | 24 |
| 015.96 | QuantSeq 3‘ mRNA-Seq Library Prep Kit (FWD) for Illumina, 96 preps | 96 |
| 015.2x96 | QuantSeq 3‘ mRNA-Seq Library Prep Kit (FWD) for Illumina, 2x96 preps | 2x96 |
| 015.384 | QuantSeq 3‘ mRNA-Seq Library Prep Kit (FWD) for Illumina HT including i5 Dual Indexing Add-on Kit (5001-5004), 384 preps | 384 |
| 016.24 | QuantSeq 3’ mRNA-Seq Library Prep Kit (REV) for Illumina with Custom Sequencing Primer, 24 preps | 24 |
| 016.96 | QuantSeq 3’ mRNA-Seq Library Prep Kit (REV) for Illumina with Custom Sequencing Primer, 96 preps | 96 |
| 113.96 | QuantSeq 3’ mRNA-Seq Library Prep Kit FWD with UDI 12 nt Set A1, (UDI12A_0001-0096), 1 rxn/UDI | 96 |
| 114.96 | QuantSeq 3’ mRNA-Seq Library Prep Kit FWD with UDI 12 nt Set B1, (UDI12B_0001-0096), 1 rxn/UDI | 96 |
| 115.384 | QuantSeq 3’ mRNA-Seq Library Prep Kit FWD with UDI 12 nt Sets A1-A4, (UDI12A_0001-0384), 1 rxn/UDI | 384 |
| 129.96 | QuantSeq 3’ mRNA-Seq Library Prep Kit FWD with UDI 12 nt Set A2, (UDI12A_0097-0192), 1 rxn/UDI | 96 |
| 130.96 | QuantSeq 3’ mRNA-Seq Library Prep Kit FWD with UDI 12 nt Set A3, (UDI12A_0193-0288), 1 rxn/UDI | 96 |
| 131.96 | QuantSeq 3’ mRNA-Seq Library Prep Kit FWD with UDI 12 nt Set A4, (UDI12A_0289-0384), 1 rxn/UDI | 96 |